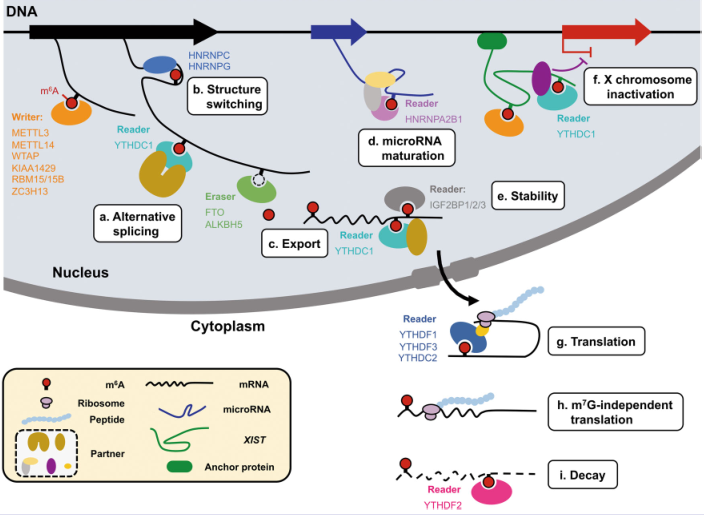
目前来说,调控m6A修饰过程的阅读蛋白共有9种功能,今天不会大家一一来讲,而是主要讲参与蛋白编码过程的YTHDF1蛋白,它主要通过与mRNA的m6A位点结合,在脑神经发育[1],多巴胺分泌[2]和突触形成[3]等过程中起重要作用。

文章导读:
2018年10月31日,美国芝加哥大学何川团队,上海科技大学周涛团队与宾夕法尼亚大学宋红军团队联合在Nature期刊发表论文,首次指出m6A 修饰对小鼠学习及记忆能力的影响,今天小编将详细对本文进行解析,同时总结一下近年来何川教授针对阅读蛋白研究的文章。
样本:野生型小鼠,YTDHF1敲除模型小鼠
技术手段:m6A RNA 甲基化测序,mRNA 测序,CLIP测序和小鼠认知相关功能实验等
时间点:初次刺激期,LTP期(刺激反应晚期)和二次刺激期
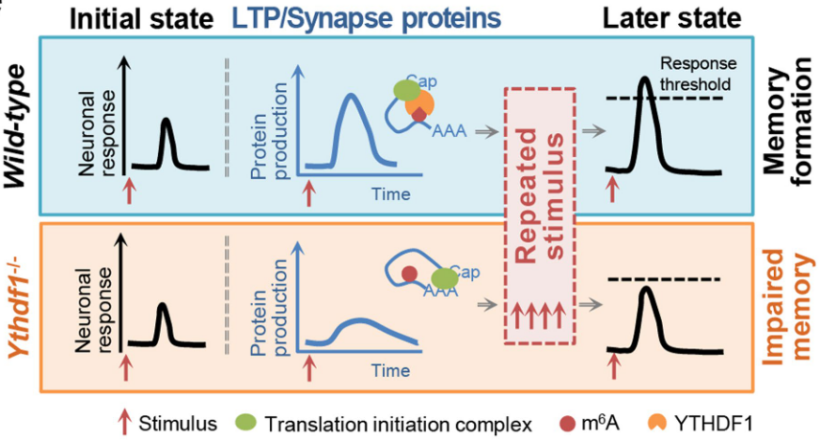
本文研究了YTHDF1敲除及野生型小鼠海马体神经受到初次刺激,LTP期和二次刺激后神经的表型反应,相关蛋白生成量及前后等电位点阈值的变化情况。
受到初次刺激时,两组小鼠对于刺激的表型反应差距不大;LTP期,YTHDF1敲除小鼠由于缺乏YTDHF1蛋白从而抑制了LTP相关蛋白的合成,导致在受到后续刺激时,等电位点变化达不到阈值影响记忆形成的能力。
1. YTDHF1敲除小鼠对空间记忆能力和外界刺激的敏感度差
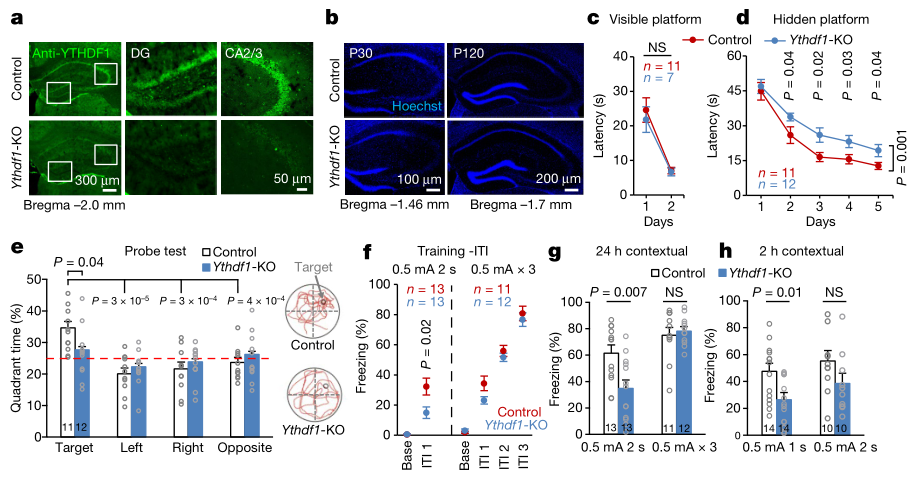
前期实验已证实YTDHF1在小鼠海马体中高表达[4],于是作者通过CRISPR-Cas9技术构建了YTDHF1敲除的模型小鼠(图1a),命名为YTDHF1-KO小鼠。与野生型小鼠相比,KO组小鼠在四周岁前,海马体的组织形态(图1b),透明水迷宫训练结果(图1c)与正常小鼠一致。而在不视水迷宫训练(图1d),探头实验(图1e)和对于外界刺激(图1f,g,h)的敏感度都要长于对照,说明敲除YTDHF1蛋白对空间记忆和外界刺激能力的反应起重要作用。
2. YTDHF1及相关蛋白参与学习和记忆关键的LTP期
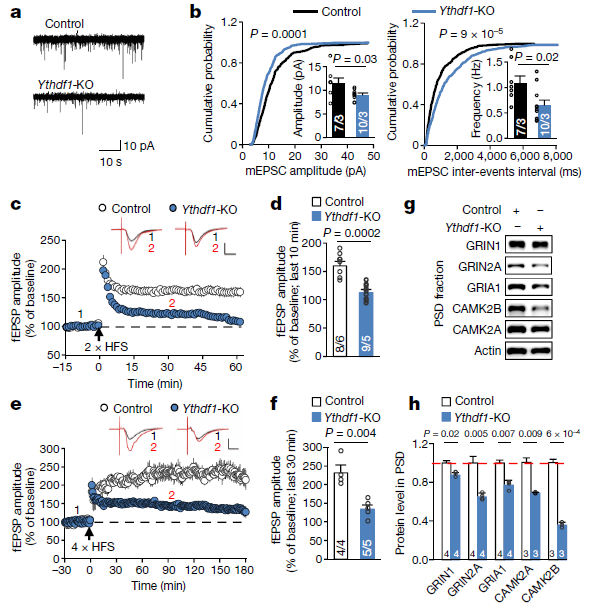
作者接着从生理水平观测敲降组小鼠脑的变化。敲降组突触电流振幅和频率比对照组低(图2 a,b),且形态比对照组小(图2 c,d)。在受到外界刺激时,神经间突触后电位阈值的变化量低于对照组(图2 e,f)。同时,此过程可能与5个与LTP期关键的突触后密度相关的蛋白相关(图2 g,h),YTDHF1回补实验显示上述表型又得到了回复。
3. YTHDF1 CLIP测序揭示m6A peak与突触传导和LTP期相关
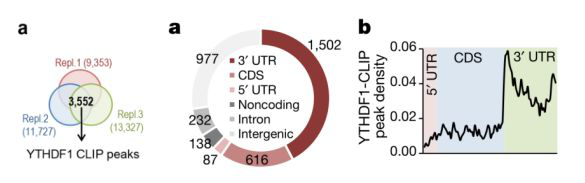
为了从分子角度进一步解析YTHDF1的作用机制,作者运用YTHDF1-CLIP测序技术,检测了小鼠海马体中(n=3,4只小鼠混为一个样本)YTHDF1 mRNA上所有的结合位点和m6A 位点。通过对三个样本的m6A peak峰取交集,在1,042个转录本上共找到了3,552个peak峰(图4a左)。1,502个peak与基因组3’UTR区匹配(图4 a右);67%的peak与转录本匹配,并在3’UTR区富集(图4 b)。对上述peaks相关的转录本进行GO和KEGG注释后,发现其与突触传导和LTP期有关,这也与之前的实验结果不谋而合。

4. m6A CLIP测序与YTHDF1 CLIP测序联合分析筛选到可能与YTHDF1结合的m6A位点
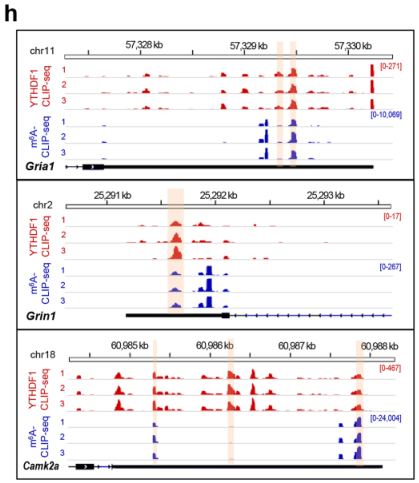
接着,作者对同类型样本进行了m6A CLIP测序,对三个样本分别进行motif分析后,发现三者共有将近有11,000个序列为GGACU的peaks(图5 c)。将peak分别于转录本和基因组比对后,发现与YTHDF1 CLIP的结果十分类似(图5 d,e)。对两次CLIP实验的peaks取交集后,发现共有将近30%的peak(图5 g),IGV结果表明:YTHDF1的m6A位点与突触形成关键蛋白(如Gira1,Grin1和Camk2a等)的m6A位点相近,二者可能通过m6A位点结合,调控蛋白的合成(图5 h)。

5. 首次绘制电休克小鼠的m6A RNA甲基化谱
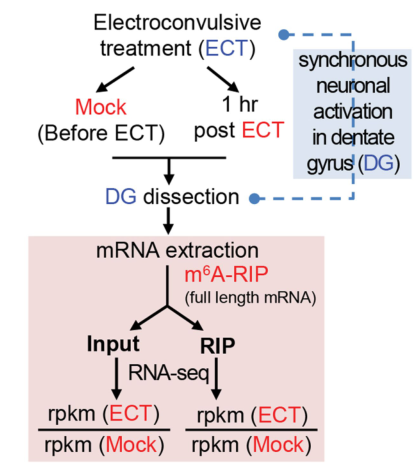
最后,作者建立了电休克小鼠模型,对海马体的齿状回结构的组织进行m6A RNA甲基化测序和转录组测序联合,快速找到许多既发生m6A 甲基化表达量又发生巨大变化的基因,以供后续研究。
总结:
本篇文章表明小鼠海马体中m6A RNA甲基化能够调控学习和记忆的生成,并且可以影响调控与神经形成相关的关键靶基因的表达,为今后进一步研究m6A修饰在学习和记忆分析机制提供了认识。
何川教授m6A RNA甲基化Reader相关蛋白成果总结:
1. 2013.11 Nature IF: 41.58
带有YTH结构域的结合蛋白通过mRNA结合影响功能。

2. 2014.5 Nature IF: 41.58
讲明Reader蛋白的作用机制。

3. 2015.2 Nautre IF: 41.58
揭示m6A 甲基化结合蛋白HNRNPC与LncRNA结合的生物学功能。

4. 2017.4 Cancer Cell IF: 22.84
揭示m6A甲基化结合蛋白HUR与LncRNA结合的生物学功能。

参考文献:
1. Hess, M. E. et al. The fat mass and obesity associated gene (Fto) regulates activity of the dopaminergic midbrain circuitry. Nat. Neurosci. 16, 1042-1048 (2013).
2. Weng, Y.-L. et al. Epitranscriptomic m 6 A regulation of axon regeneration in the adult mammalian nervous system. Neuron 97, 313-325.e6 (2018).
3. Li, L. et al. Fat mass and obesity-associated (FTO) protein regulates adult neurogenesis. Hum. Mol. Genet. 26, 2398-2411 (2017).
4. Lein, E. S. et al. Genome-wide atlas of gene expression in the adult mouse brain. Nature 445, 168-176 (2007).
云序生物国内独家提供RNA甲基化测序一站式服务
云序生物提供比色法检测整体m6A甲基化修饰水平、RNA甲基化测序、MeRIP-PCR验证和RIP,RNA Pull Down机制研究服务。RNA甲基化测序技术是真正实现m6A,m5C和m1A修饰,检测分子除mRNA外,还能检测环状RNA,LncRNA及其他非编码RNA。2016年至今,样本数量累积超过1000+,MeRIP富集成功率高达98%以上。
现在,为解决客户样本量少的问题,特推出超微量MeRIP测序技术,500ng总RNA既可进行测序实验。特殊样本也可进行RNA甲基化测序,如血清,血浆,外泌体和石蜡样本。
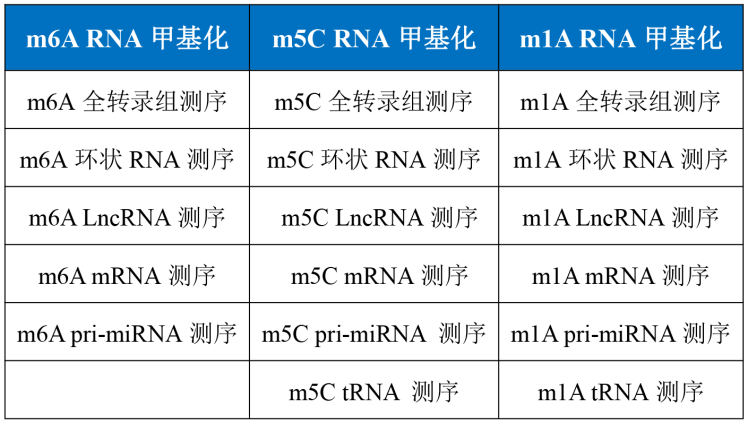
云序生物RNA甲基化产品列表:
产品链接:
RNA Pull Down
上海云序生物科技有限公司
Shanghai Cloud-seq Biotech Co., Ltd.
地址:上海市松江区莘砖公路518号20号楼3楼
电话:021-64878766
传真:021-64878766
网址:www.cloud-seq.com.cn
邮箱:market@cloud-seq.com.cn
